Cómo los cantos de los saltamontes
amplían los conocimientos de los
investigadores sobre los insectos
tropicales
Visualice
lo invisible
Una comunidad de científicos rearmó los ecosistemas microbianos por medio de anvi’o
Jarrod J. Scott, becario postdoctoral, es un colaborador activo de anvi’o, un conjunto de herramientas informáticas para visualizar comunidades microbianas.
Si una mano mágica pudiera pesar toda la vida del océano, los microbios representarían el noventa por ciento de la biomasa total. Nuestra salud y la salud del medio ambiente dependen de comunidades de vida demasiado pequeñas para verlas. Una sola gota de agua de mar puede contener 10 millones de virus y un millón de bacterias. Más de 1.5 billones de bacterias habitan la piel de una persona. Los investigadores del Smithsonian están utilizando una infraestructura computacional y de visualización avanzada para comprender estos pequeños universos en los ecosistemas con mayor biodiversidad del mundo.
Jarrod J. Scott, becario postdoctoral patrocinado por la Fundación Moore en el Instituto Smithsonian de Investigaciones Tropicales (STRI) junto a sus colegas, son como médicos en una sala de emergencias que necesitan saber con urgencia si un arrecife de coral está sano o enfermo. Para investigar los microorganismos que viven en distintos entornos, trabajan con un equipo inteligente, dirigido por A. Murat Eren (Meren), profesor asistente de la Universidad de Chicago, que desarrolló un conjunto de herramientas completamente de código abierto e impulsado por la comunidad llamado anvi’o para procesar, analizar y visualizar datos ómicos de comunidades microbianas.
A. Murat Eren (Meren), profesor asistente de la Universidad de Chicago durante un viaje de campo para los participantes en el taller Microbioma marino 2019 de STRI. Crédito: Jorge Alemán.
"Bajo una montaña de datos, los microbiólogos pueden tener dificultades para ver patrones", comentó Jarrod Scott. “Los seres humanos son buenos para identificar patrones porque los patrones contienen información útil. Anvi'o funciona como nuestros ojos en el mundo microbiano. Un biólogo puede contarle mucho a partir de una imagen de un bosque; no necesitan complicarlo con un montón de nombres en latín o hablando de hojas pinnadas compuestas. Anvi'o es nuestra forma de crear imágenes de sistemas microbianos complejos".
Anvi’o, que significa Análisis y Visualización de Datos Ómicos (Analysis and Visualization of ‘Omics Data), consta de más de 100,000 líneas de código informático que ayuda a los investigadores a reconstruir comunidades microbianas a partir de secuencias de ADN. Para comprender cómo funciona, pruebe este rompecabezas. Coloquemos las siguientes frases en un orden que tenga sentido:
- “20 minutos. Combine el arroz con el vinagre, el aceite, el azúcar y la sal”.
- “como los vio Don Quijote, comentó a su escudero: 'La fortuna está guiando nuestros asuntos mejor de lo que hubiéramos esperado. Mira"
- “cacerola combinar con agua. Llevar a ebullición, bajar el fuego y cocinar por"
- “Enjuague el arroz en un colador hasta que el agua salga clara. En media"
- “En ese momento, descubrieron treinta o cuarenta molinos de viento en esa llanura. Tan pronto"
- “allá, Sancho Panza, amigo mío, donde hay treinta o más gigantes monstruosos con los que pienso pelear”.
Las frases se clasifican en dos conjuntos o contenedores: El contenedor de "instrucciones de cocción" contiene las frases 1, 3 y 4. El contenedor de "Don Quijote" contiene las frases 2, 5 y 6. Según la estructura de la oración y el orden de las palabras, es fácil de poner las frases en orden: “Instrucciones de cocina”, 4, 3, 1 y “Don Quijote”, 5, 2, 6 para crear historias significativas. Bastante sencillo, ¿verdad?
Ahora imagine la enorme tarea de clasificar millones de frases como las de arriba de una enorme biblioteca de libros triturados. Y su objetivo es volver a armar todos los libros. Esta situación es análoga a la que enfrentan los ecologistas microbianos cuando estudian microbios ambientales. Pero en lugar de clasificar palabras y oraciones en libros, los ecologistas microbianos necesitan clasificar secuencias de ADN y genes en genomas. El medio ambiente es la biblioteca y los genomas microbianos son los libros. Verá, para estudiar estos microbios, las células deben abrirse para liberar el ADN. En el proceso, las hebras de ADN se rompen en pequeños trozos y todos se mezclan (una enorme biblioteca de genomas triturados) y el objetivo es volver a unir el genoma.
Las secuencias de ADN constan de cadenas de pares de bases (A, G, C y T) que codifican genes. Los genes, a su vez, se traducen en secuencias de aminoácidos que dan lugar a proteínas, las biomoléculas que las células necesitan para vivir. La colección completa de genes comprende el genoma de un organismo.
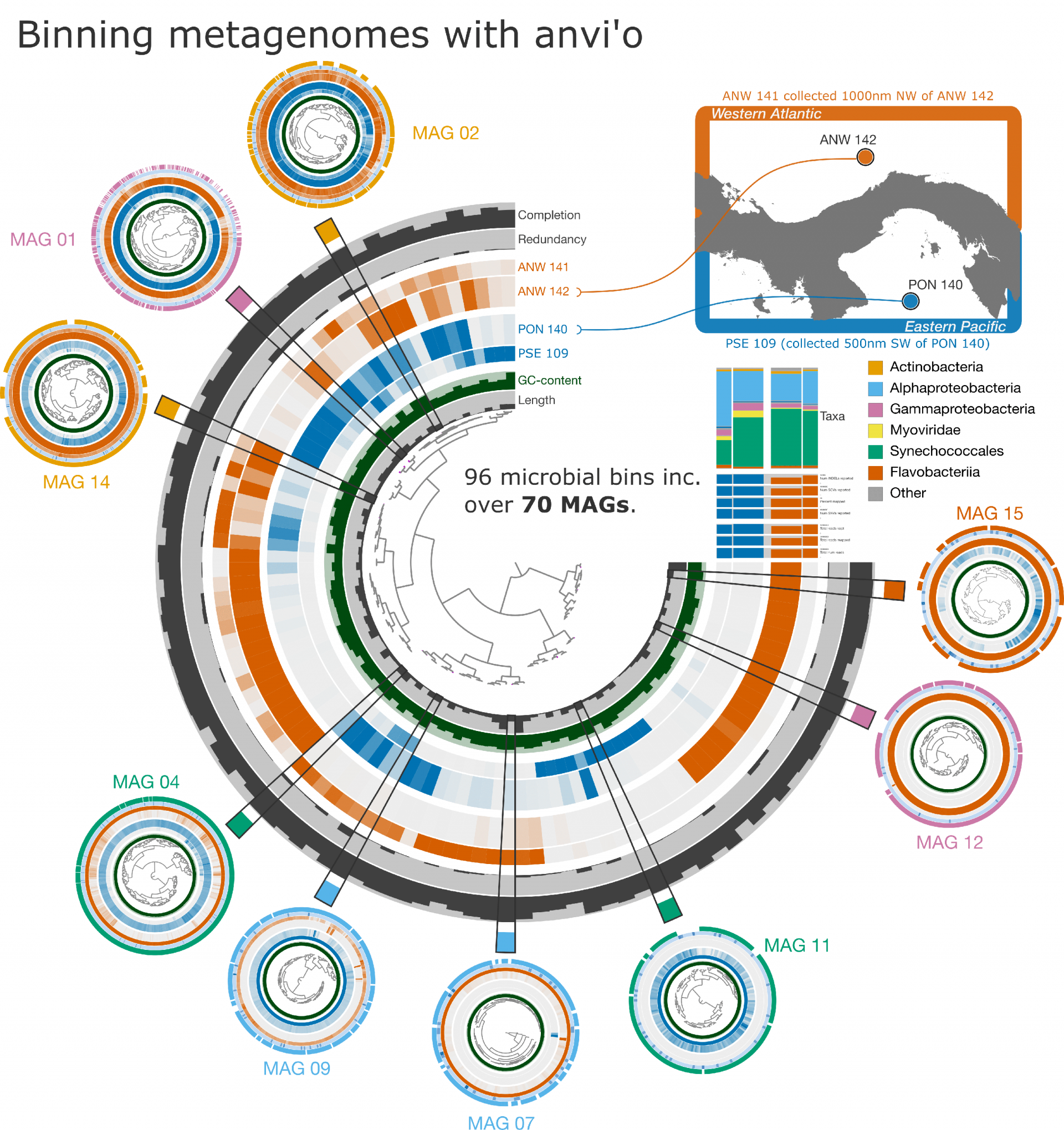
Cada anillo (imagen central) representa una muestra de metagenoma, codificada con colores por el océano donde se tomaron muestras de microbios. Los radios dentro del anillo indican distintos contenedores microbianos generados por el ensamblaje conjunto de los datos metagenómicos. La intensidad del color denota la abundancia de contenedores dentro de una muestra. El objetivo del co-ensamblaje y agrupamiento es reconstruir genomas microbianos en genomas ensamblados por metagenoma, o MAG. Los MAG de ejemplo se muestran en los círculos más pequeños. Para obtener una descripción de los datos utilizados en estos análisis, consulte la nota al final del artículo.
“Una cosa que hacemos cuando intentamos reconstruir genomas es buscar genes de mantenimiento, un subconjunto de genes que tienen la mayoría de las bacterias. Cuantos más de estos encontremos en un contenedor, más seguros estamos de que tenemos un genoma completo”, comentó Jarrod Scott. “Siguiendo la analogía del libro, es como si tuviéramos la página del título, la página con los derechos de autor, índice, texto principal, un conjunto de referencias y una tabla de contenido, sabemos que tenemos el libro completo”.
Una frase, o secuencia, de código de ADN de un entorno podría provenir de cualquiera de miles de organismos diferentes: ballenas y tiburones hasta bacterias y virus. En nuestro pequeño rompecabezas anterior, usamos contenido y contexto para agrupar y ordenar las oraciones fragmentadas para crear historias significativas. Al comparar las secuencias de ADN entre sí y con bases de datos de organismos conocidos, los investigadores pueden agrupar estos fragmentos de ADN en distintos contenedores; el contenedor de ballenas, el contenedor de tiburones y contenedores que representan diferentes hongos, bacterias y virus. Es más fácil decirlo que hacerlo, y aquí es donde anvi'o ha revolucionado el campo de la ecología microbiana.
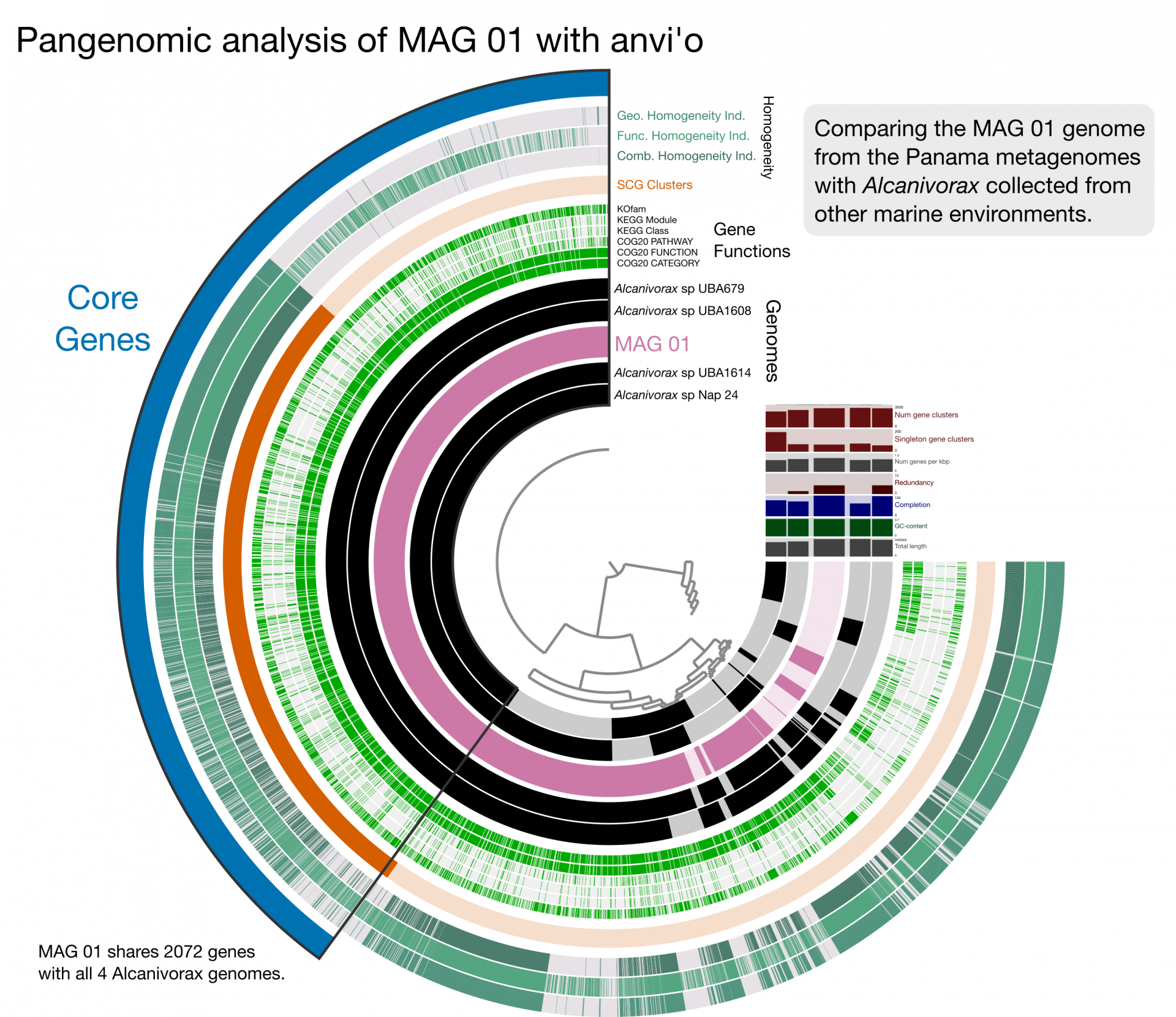
Para determinar qué genes comparten todos los genomas y qué genes pueden ser específicos de un hábitat o entorno en particular, los investigadores inician comparando las secuencias de ADN en sus muestras con secuencias en grandes bases de datos que contienen secuencias conocidas de ADN de muchos organismos distintos para determinar la identificación de género y especie de cada genoma metaensamblado o MAG. MAG-01, abundante en ambos océanos, puede representar a Alcanivorax, un microbio marino común. Aquí, se utiliza un análisis pangenómico para comparar MAG-01 con genomas relacionados de bases de datos públicas. En este caso, cada anillo representa genomas y los radios representan grupos de genes. El color indica la presencia de un grupo de genes.
Los autores de un nuevo artículo sobre la historia, la evolución y el desarrollo de la comunidad de anvi’o en Nature Microbiology utilizan una analogía de cocina para describir la forma en que el software permite al chef, en este caso un microbiólogo, crear imágenes visuales de datos. El software permite a los investigadores intercambiar diferentes flujos de trabajo modulares (mezclar todos los ingredientes secos y luego agregar los ingredientes húmedos) para ver cómo cambia el resultado. Anvi’o también brinda a las personas la libertad artística de cambiar los colores, las etiquetas y el orden de la información que presentan para crear representaciones claramente comprensibles (consulte las imágenes de esta historia para ver ejemplos).
Anvi'o es un ecosistema de más de 140 herramientas interconectadas que ayuda a los investigadores a desentrañar la diversidad microbiana. Como le gusta decir a Meren, "anvi'o te permite ensuciarte las manos". Ofrece a los usuarios una experiencia mejorada y la mayor libertad analítica posible, incluidas amplias capacidades de visualización interactiva que permiten a los usuarios explorar estos datos de formas que antes no eran posibles.
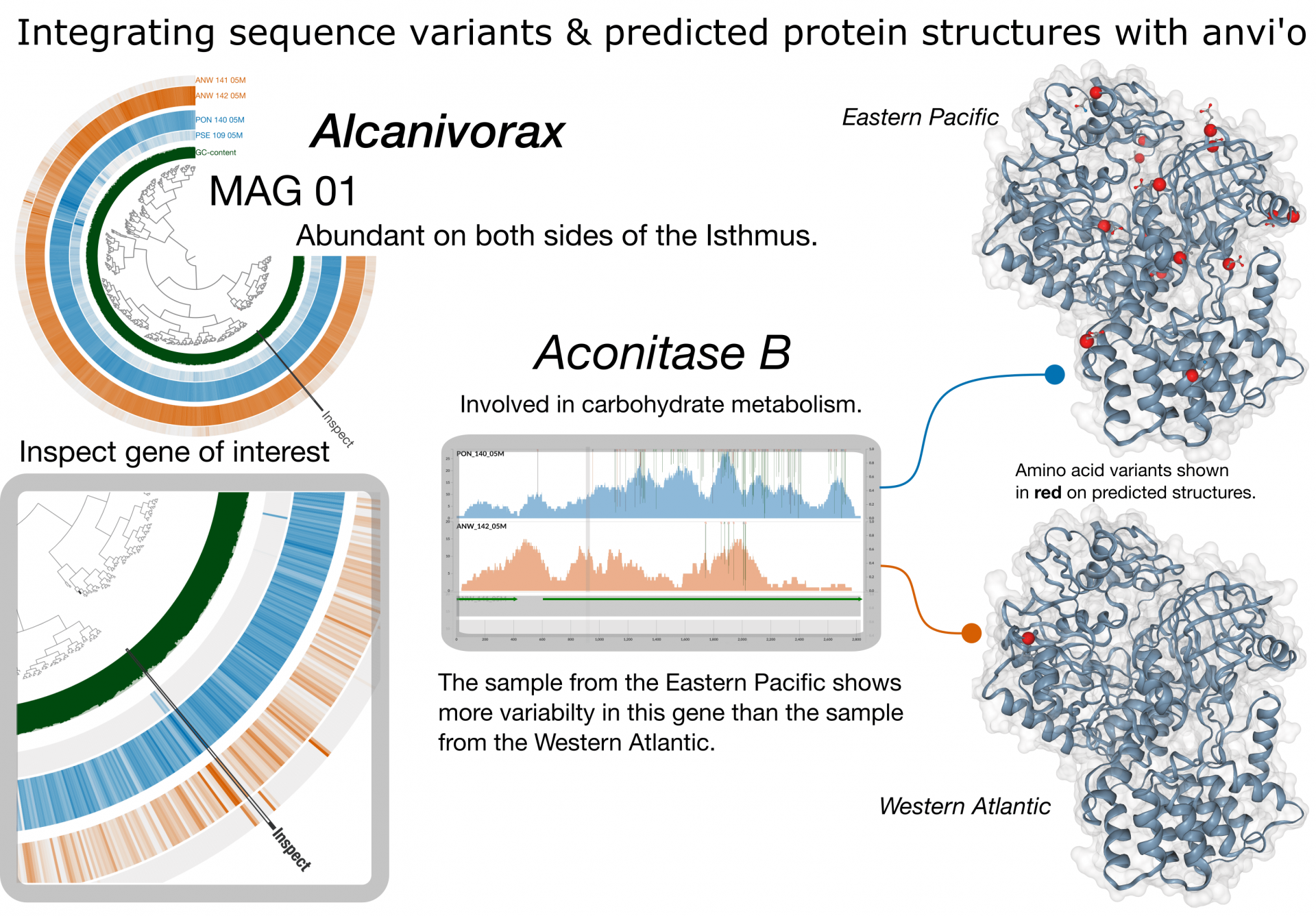
Aunque MAG-01 es abundante en ambos océanos, el Pacífico Oriental y el Atlántico Occidental difieren dramáticamente en sus propiedades geoquímicas y físicas. Una forma de comprender cómo el entorno puede moldear la evolución del genoma es inspeccionar los perfiles de variabilidad a nivel de genes (por ejemplo, codones o aminoácidos). El perfil que se muestra aquí indica una mayor variabilidad en el metagenoma del Pacífico Oriental. Los residuos variables se mapean luego en la estructura predicada de la proteína resultante. Los cambios en la secuencia de aminoácidos pueden alterar la forma de una proteína y dichos cambios pueden influir en la función de la proteína.
Una comunidad de científicos cada vez más amplia contribuye a mejorar a anvi'o como herramienta: personas que estudian virus, informática, astrobiología, microbios en aguas termales, genes de resistencia a los antibióticos ¡e incluso el genoma tardígrado! Las versiones del programa se nombran como un pequeño tributo a importantes mujeres científicas; la versión actual lleva el nombre de Hope E. Hopps, especialista en enfermedades infecciosas que junto con sus colegas desarrollaron una vacuna para la rubéola.
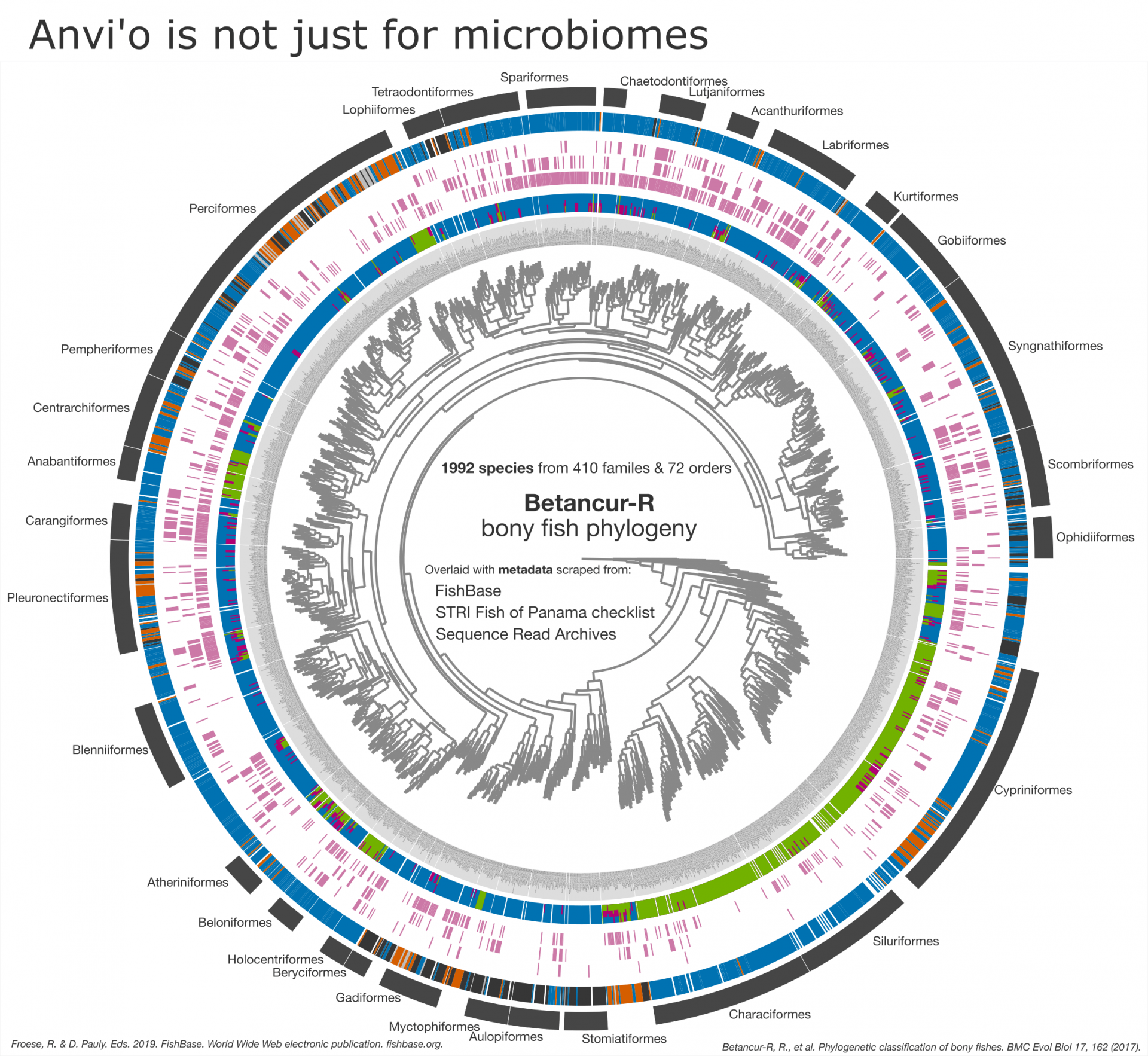
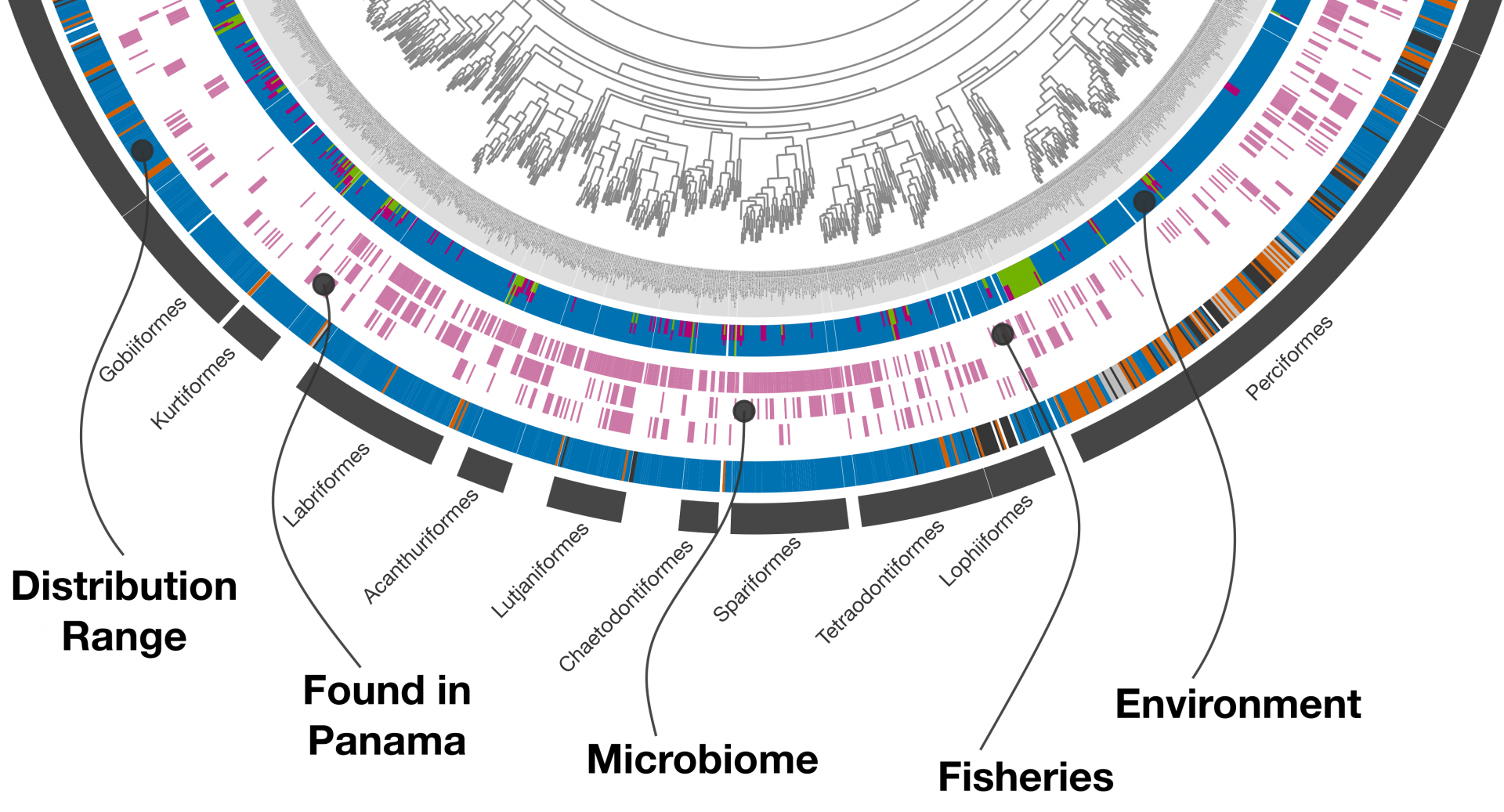
Jarrod Scott se encuentra entre los más de veinte autores del artículo porque es un colaborador activo de la comunidad anvi’o. Conoció a Meren en el 2015 y ahora trabaja en el Smithsonian en Panamá en un gran proyecto que utiliza anvi'o para comprender cómo la hipoxia aguda (bajo nivel de oxígeno) afecta los ecosistemas marinos cercanos a la costa. También está usando esta herramienta para comparar comunidades microbianas marinas de las áreas del Atlántico occidental y Pacífico oriental de Panamá. Scott incluso está creando una filogenia interactiva de peces óseos en anvi'o utilizando un árbol previamente publicado (Betancur-R et. Al., 2017) y datos que extrajo de la plataforma FishBase para cada especie. "Esa es la belleza de anvi'o", comentó Scott. “No es solo para análisis microbianos, puede usarse para muchos tipos de datos distintos”.
El análisis metagenómico se encuentra en el núcleo de anvi’o, pero gracias a sus amplias capacidades de visualización, Anvi’o se puede utilizar para muchos otros tipos de datos. Aquí, una filogenia de peces óseos publicada anteriormente por Betancur-R et. al. (2017) es combinada con metadata extraída de FishBase, Sequence Read Archive (SRA) de NCBI y la lista de verificación de STRI sobre los Peces de Panamá.
"No se equivoque, anvi’o tiene una curva de aprendizaje empinada, pero vale la pena las lágrimas que derramará" Jarrod Scott comentó: “Los miembros del laboratorio de Meren y los colaboradores externos han escrito extensos tutoriales que hacen que el aprendizaje sea más accesible y divertido. De hecho, recomendaría estos recursos a cualquiera que trabaje con "datos ómicos, ya sean expertos o recién llegados al campo". El laboratorio de Meren también mantiene un sitio de anvi’o Slack muy activo con más de 700 miembros donde las personas pueden obtener ayuda de la comunidad.
La búsqueda por web dio como resultado más de 40 piezas de propiedades de metadatos a nivel de especie. Por simplicidad, solo se muestran 5. Comenzando en el anillo más externo, el rango de distribución indica dónde se encuentra la especie (tropical, templado, de aguas profundas, etc.). Encontrado en Panamá indica si se han registrado peces en algún lugar de Panamá. El microbioma (solo a nivel de género de peces) indica si se encontraron datos de secuencia microbiana en el SRA. Pesquerías contiene información sobre si un pez se utiliza como alimento, desde pesquerías altamente comerciales hasta de subsistencia. El medioambiente describe si la especie vive en sistemas de agua dulce, salobre y/o marinos.
Una nota sobre los datos microbianos utilizados en los ejemplos anteriores: Entre el 2009 y 2013, se recogió ADN de bacterias, virus y otros microbios en más de 200 estaciones de muestreo de todos los océanos del mundo durante la Expedición global de Tara Oceans (Sunagawa et. al., 2015). La expedición recolectó ADN microbiano de sitios en el Atlántico Occidental de Panamá cerca de la ciudad portuaria de Colón y del Pacífico Oriental en la Bahía de Panamá. En este artículo, analizamos un pequeño subconjunto de estas muestras usando anvi’o para demostrar cómo nos ayuda a visualizar y explorar comunidades microbianas. Tenga en cuenta que este análisis es solo para fines de demostración. Para un análisis más riguroso y completo de los microbiomas de Tara Oceans, consulte la increíble publicación del 2018 de Delmont y sus colegas.
Eren, A.M., Kiefl, E., Shaiber, A. et al. Community-led, integrated, reproducible multi-omics with anvi’o. Nat Microbiol 6, 3–6 (2021). https://doi.org/10.1038/s41564-020-00834-3